现在UK Biobank项目申请Proposal已经成功获批后,只能通过UKB-RAP平台访问与下载。UKB目前与AMS和DNAnexus合作,建立了云计算平台Research Analysis Platform(简称RAP,ukbiobank.dnanexus.com/),所有项目申请获批的研究人员都可以直接使用。可以下载数据到自己电脑上分析,也可以在UKB-RAP平台上的JupyterLab和Rstudio等应用中分析。
1.UKB-RAP平台基础知识
1.课题组购买数据后,申请项目成功后,加入你们的项目组,获取ukb个人账户。
2.成功注册UKB-RAP平台账号(https://ukbiobank.dnanexus.com),注册时有可能因为网络问题,无法加载出人机验证界面,可适当使用魔法。
3.进入RAP平台,创建Project,填写上UKB账户里面的 Application ID。注意勾选上Dispense bulk data files... ,其包含了基因数据,影像数据和一些体力活动数据,说不定后续研究就用上了呢。
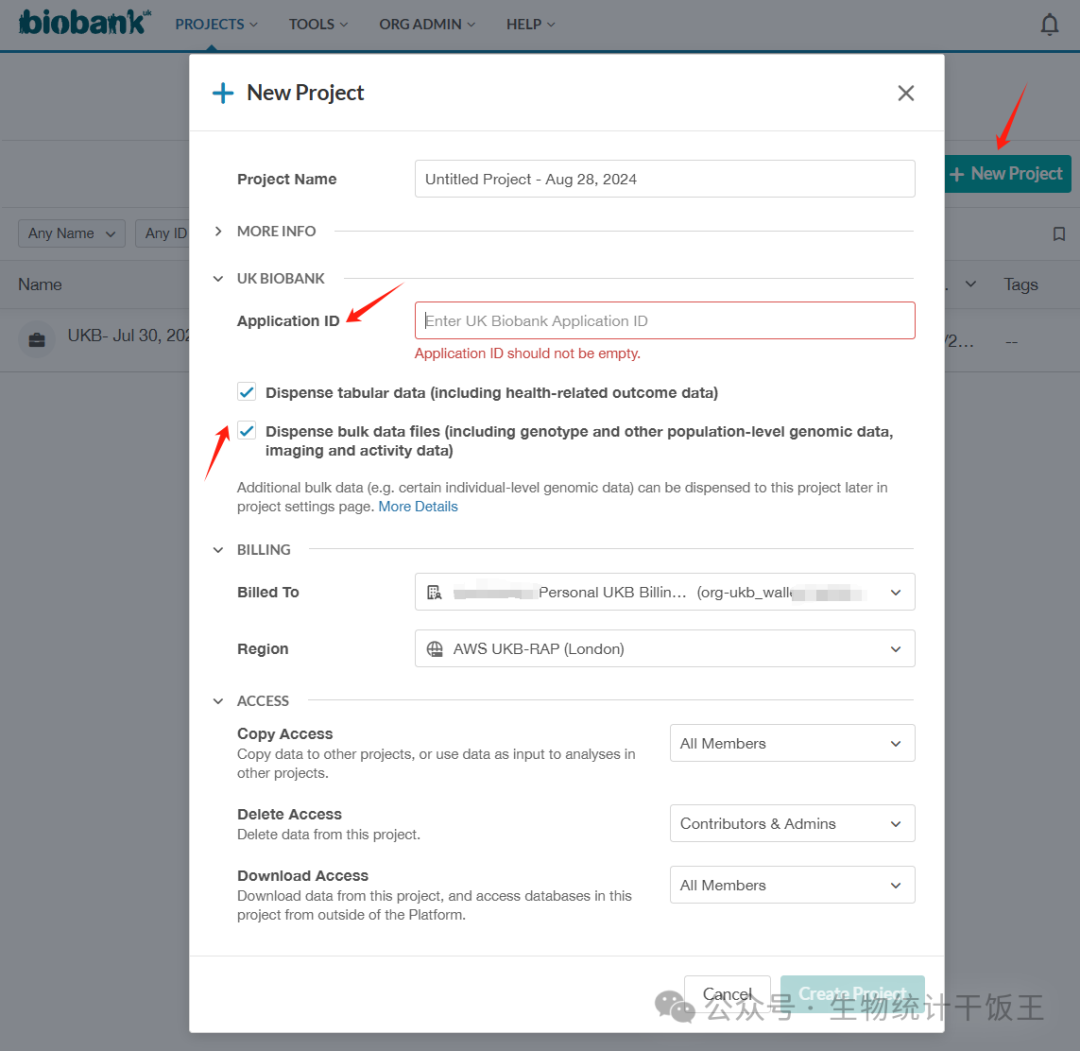
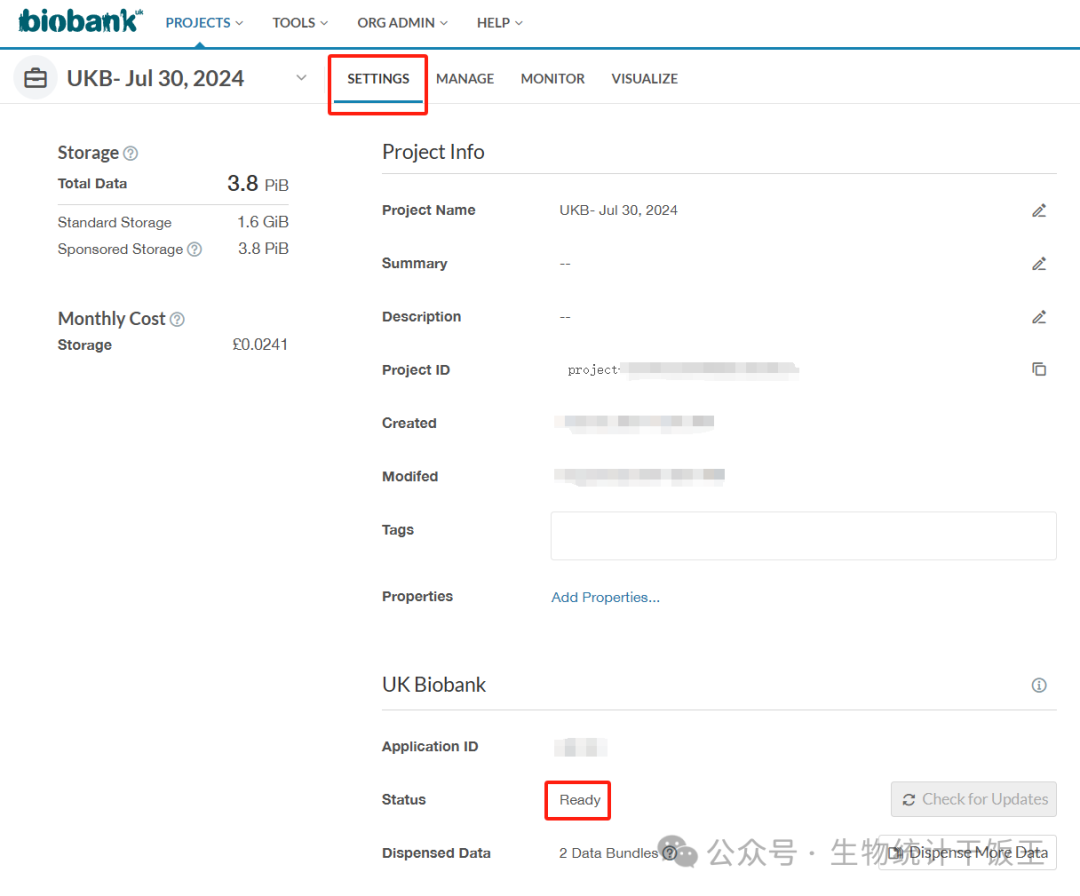
4.创建成功后,等待ukb分发数据到你的项目里,大概1天左右时间吧。在SETTINGS界面里面查看状态刚开始是 Dispensing data,等待个1天左右,变为 Ready 时就可以使用了。
5.在MANAGE界面中得到数据的项目文件夹如下,绿框中是ukb给我们分发的数据,不用去改动其中的文件。可以自己创建个test文件夹(红框中)来保存提取和分析产生的数据。例如,可以将使用RAP平台上的Table exporter,JupyterLab或Rstudio等应用产生的数据上传到其中。
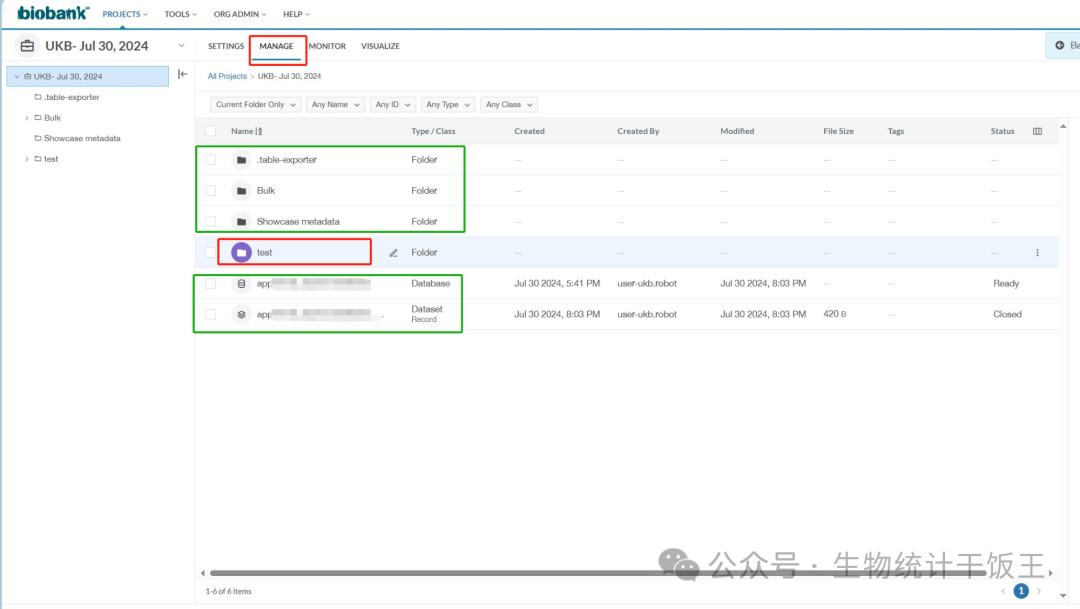
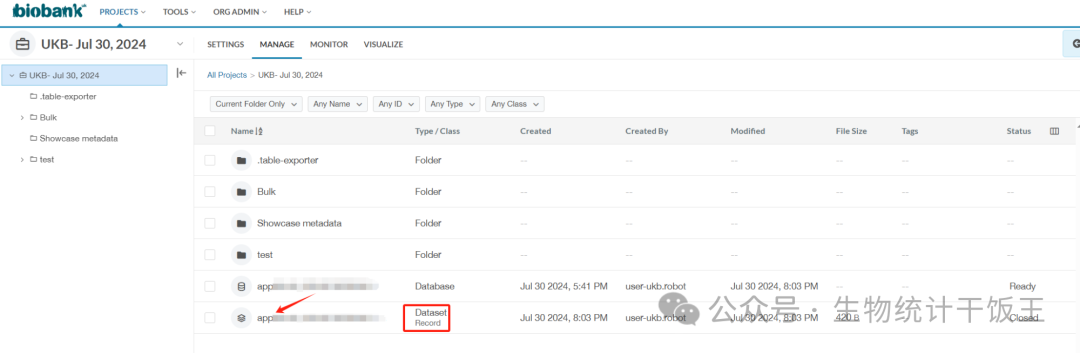
- 类型为Dataset的app...开头的文件(下图的最后一个),即是一般数据。如Population characteristics、Assessment centre、Biological samples、Health-related outcomes等文件夹里面展示的变量(变量详情见 https://biobank.ctsu.ox.ac.uk/crystal/browse.cgi?id=2&cd=browse)。
- bulk文件夹包含了基因数据,影像数据和一些体力活动数据,无法直接下载。不过可以通过JupyterLab、Rstudio或Swiss Army Knife应用分析处理后,下载处理后的结果。
2.使用Table exporter下载一般数据
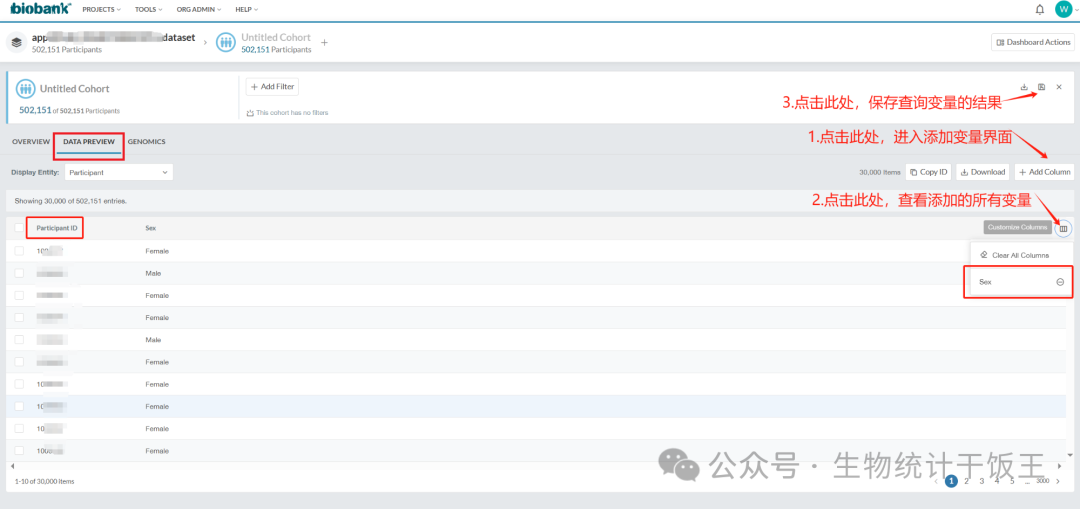
1.进入数据库,添加需要的变量
- 进入DATA PREVIEW界面,其中自带Participant ID变量,这里展示添加一个Sex变量。添加的变量超过30个,就无法预览了,可以点击下图中的2,查看添加的所有变量。
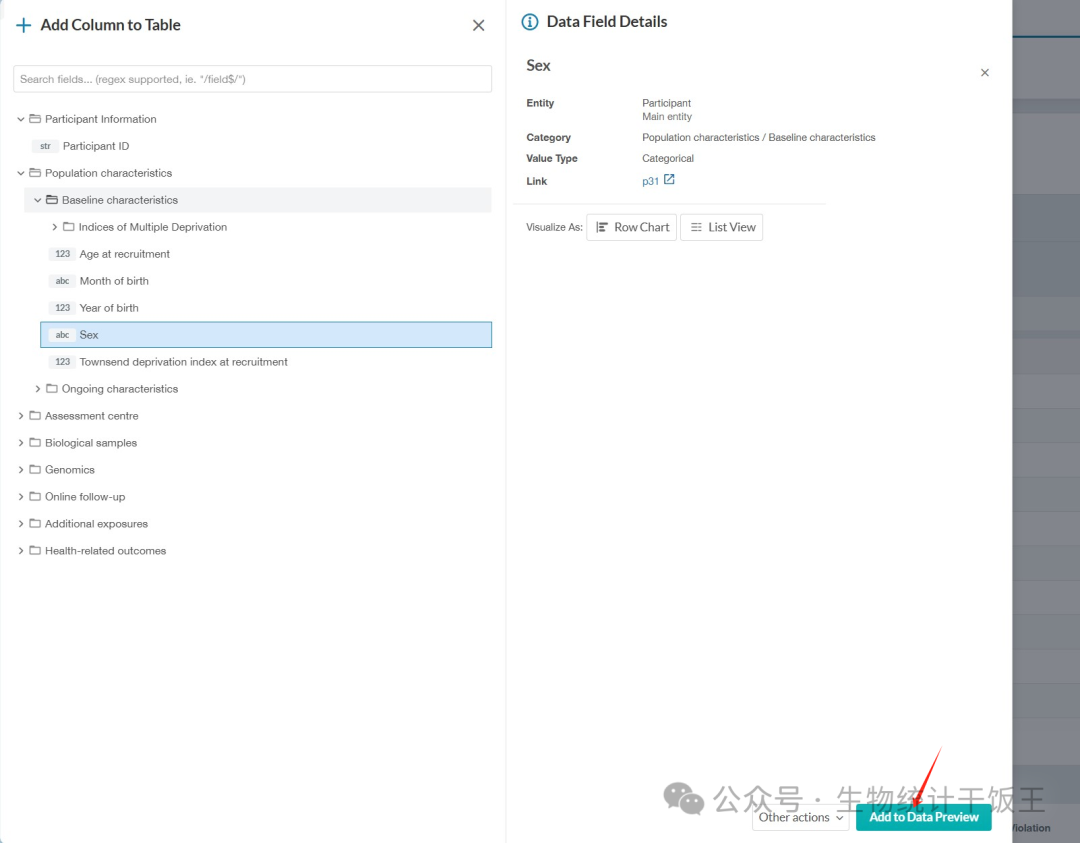
- 在变量添加界面(变量详情见 https://biobank.ctsu.ox.ac.uk/crystal/browse.cgi?id=2&cd=browse),找到Sex变量,点击 Add to Data Preview,添加成功后变为 Column Added。
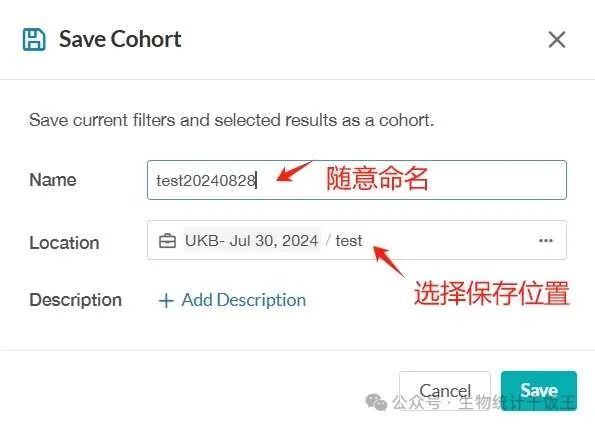
- 接下来点击保存查询到的变量,保存到个人的文件夹,这里选择前文建立的test文件夹。
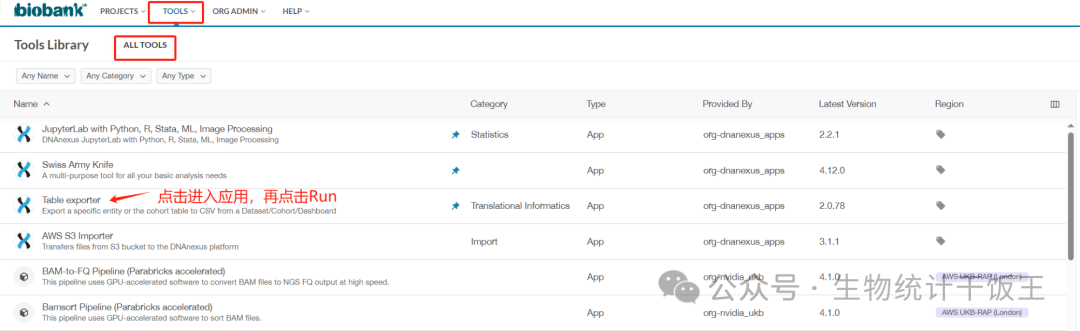
2.使用Table exporter应用将查询转换为csv文件,然后下载
- 在Tools中的Tools Library中,找到Table exporter应用,点击进入后,出现介绍页面,直接点击绿色按钮Run。
- 运行Table exporter应用,选择输出位置。
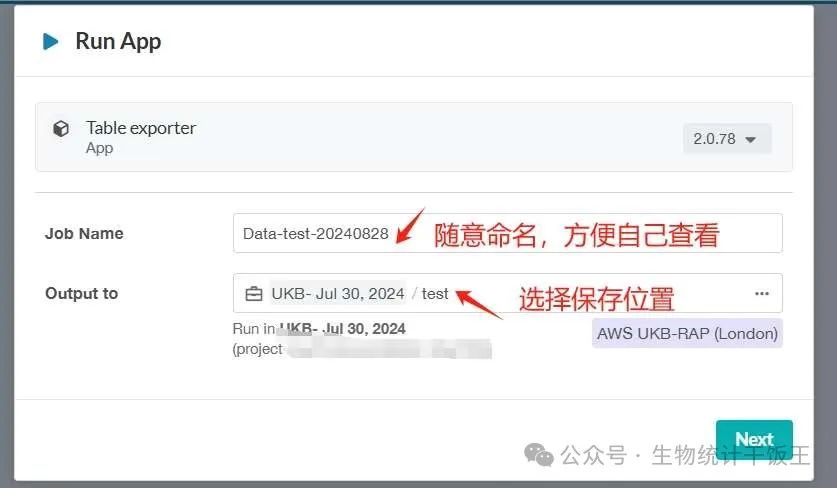
- Table exporter运行前的设置,设置好后,直接开始分析。
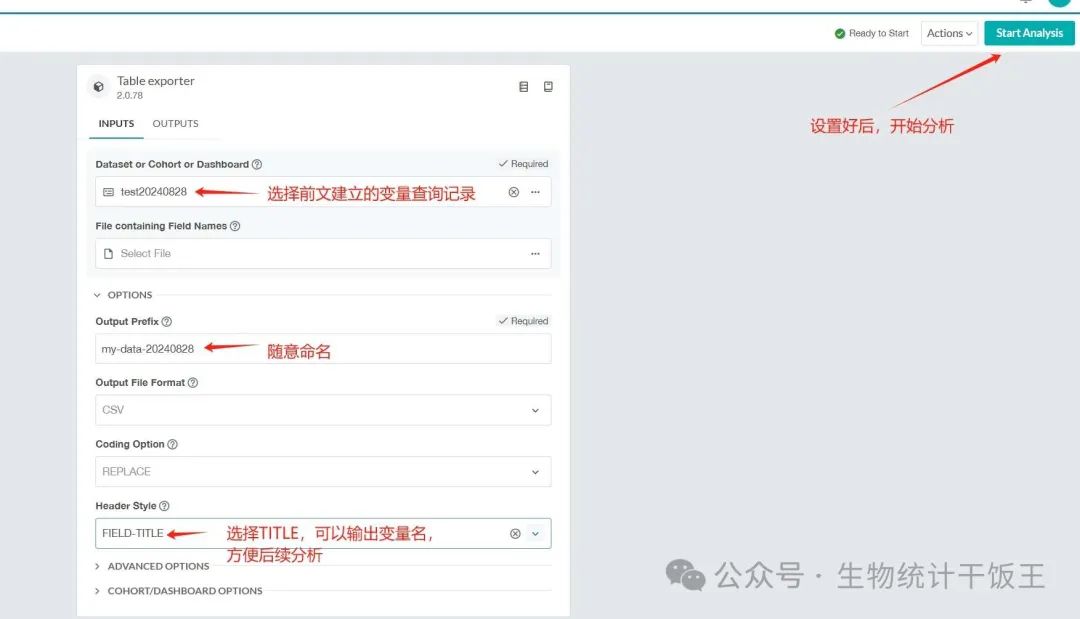
- 确认开始分析的界面,可以选择设备配置,然后运行。
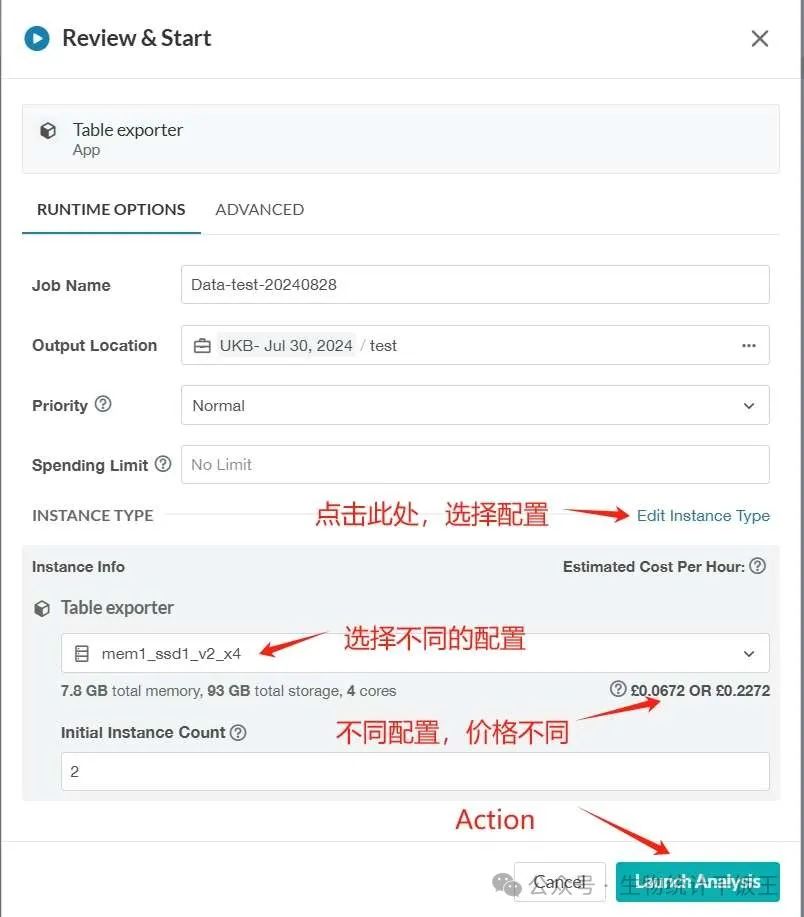
- 运行时可以在项目的 MONITOR 界面,查看运行状态,该开始可能是Waiting,等一会就会开始运行了。运行花费会实时更新,转换数据成本很低,一般不超过0.1。运行Done后,就可以得到csv文件了。
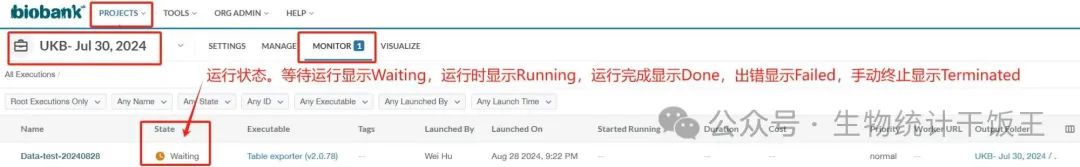
- 点击csv文件,然后下载,下载速度很快,经过小编测试一般2M/s以上。然后就可以愉快的在自己电脑上分析数据了。

3. 使用Swiss Army Knife应用提取SNP数据
如果我们想要提取某些SNP另作他用,此时就需要Swiss Army Knife应用,其包含了众多生物信息学工具,如plink可用于提取Bulk/Genotype Results/Genotype calls文件夹中的未插补的基因数据中的SNP信息,BGEN可用于提取Bulk/Imputation/UKB imputation from genotype文件夹下插补后的基因数据中的SNP信息。
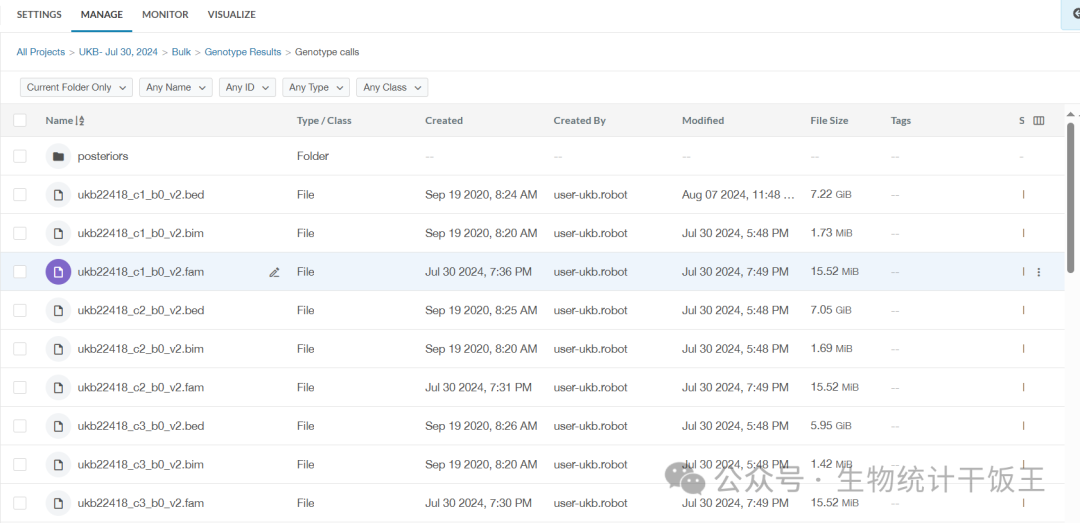
这里我们简单演示如何提取Genotype calls文件夹下的SNP数据,文件是pink的二进制格式,每条染色体是分开的,详情如下图所示。
步骤1. 2. 点击 Tools Library 中的 Swiss Army Knife应用
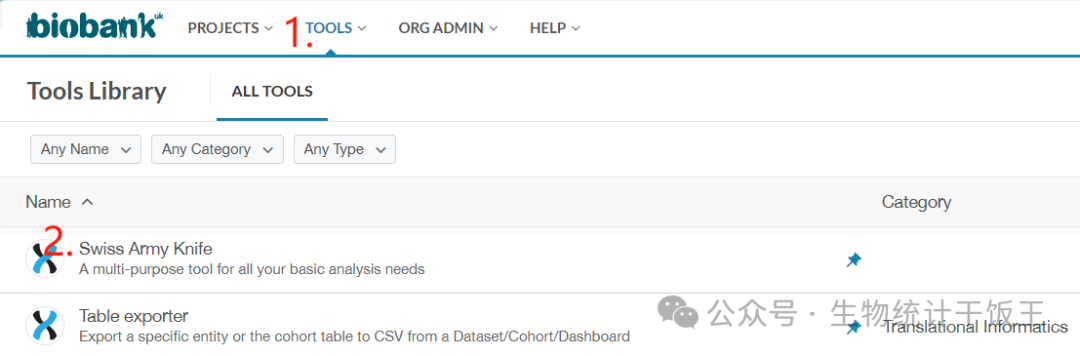
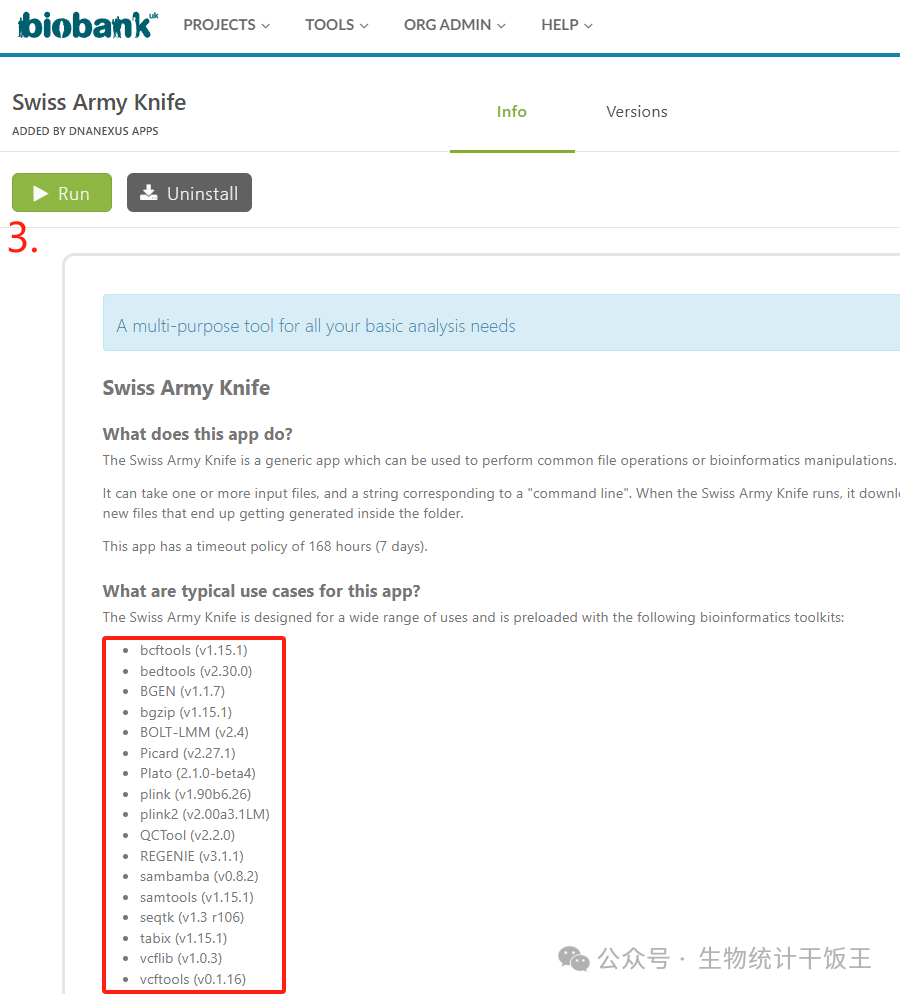
步骤3. 运行 Run, Swiss Army Knife中可用的生物信息学工具如下:https://ukbiobank.dnanexus.com/app/swiss-army-knife
步骤5. 选择输入文件,Bulk/Genotype Results/Genotype calls文件夹中,选择1-4号染色体文件,IVs.txt和 ukbtest.sh脚本,.txt文件和 .sh脚本文件在本地编辑好,然后上传到项目中。
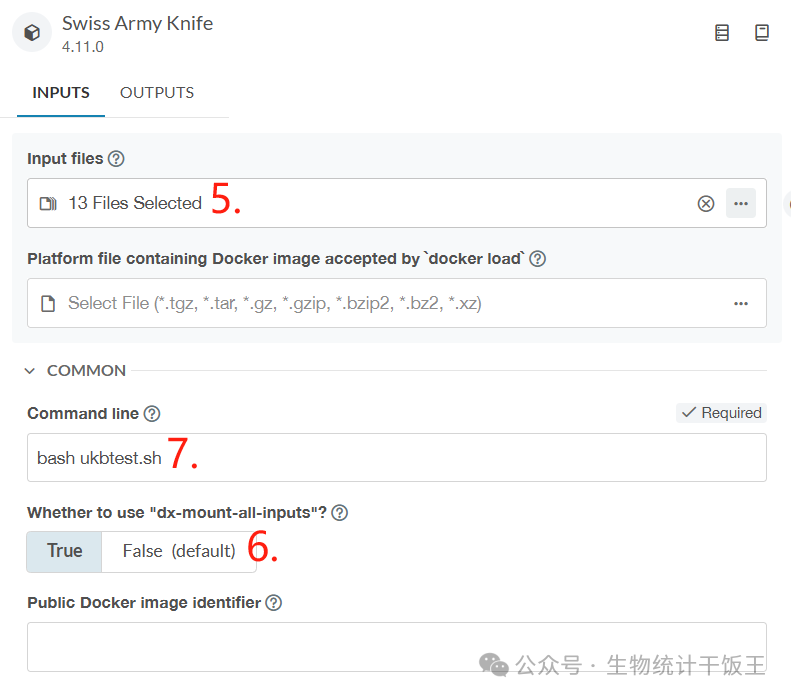
步骤6. dx-mount-all-inputs 选择 TRUE:挂载,流式传输文件不需要下载到磁盘。两种文件输入方式,直接下载 或 挂载(mount),详情:(https://dnanexus.gitbook.io/uk-biobank-rap/working-on-the-research-analysis-platform/working-with-bulk-data-files)

步骤7. 命令行输入(Command line):输入需要运行的命令,跟plink软件中一样,不了解的同学可以学习一下plink(https://www.cog-genomics.org/plink/1.9/)。这里按照包含步骤5. 6. 7.的图操作,然后点击右上角的开始分析就可以了。输入运行命令有如下两种形式:
- 直接输入命令(不太方便),如提取一个SNP rs28659788:
plink --bfile ukb22418_c1_b0_v2 --extract rs28659788 --make-bed --out test1 - 运行脚本(脚本中可包含多行命令,更方便):
bash ukbtest.sh。- ukbtest.sh脚本中的内容如下:
#!/bin/bash
#test 2. Extracting rs10172629 from chromosome 1
plink --bfile ukb22418_c1_b0_v2 --extract rs28659788 --make-bed --out test2
#test 3. Run your command across all chromosomes
for chr in {1..22}; do \
plink --bfile ukb22418_c${chr}_b0_v2 \
--extract IVs.txt \
--make-bed \
--out test3.chr${chr} ;
done- IVs.txt的内容如下(输入所有你需要的SNP的rsID,不用包含列名header):
rs10172629
rs17257408
rs6834707
# 以下可添加更多的SNP,一行一个SNP(txt文件中不要包括本行)运行结束后可以得到test2.bed,test2.bim,test2.fam,test3.bed,test3.bim,test3.fam,及.log plink运行日志文件。
以上就是本期关于UKB数据库使用的内容,更多UKB-RAP平台上其他应用的使用介绍,后续再见。
感谢西安交通大学公共卫生学院 陈方尧老师团队杨嵛惠、胡维维和陈诗宇同学撰写原文发布于《生物统计干饭王》公众号上并许可转载。


 陕公网安备61011302000964号
陕公网安备61011302000964号 扫码关注公众号
扫码关注公众号